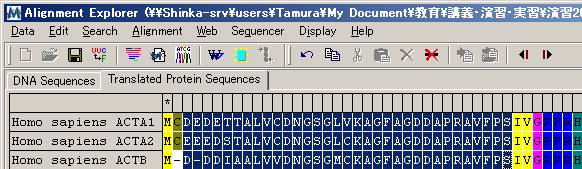
配列データのアライメント
例題データ: Actin gene coding region
(1)
「Translated Protein Sequences」タブをクリックし、アミノ酸配列を表示する。
(2)
「Ctrl+A」で全配列の全領域を選択し、ClustalW(W)ボタンをクリックする。
(3)
表示される「ClustalW Parameters」ダイアログボックス(左下図)の「OK」ボタンをクリックし、アライメントを行う。(DNA配列の場合、右下図のダイアログボックスが表示される。)
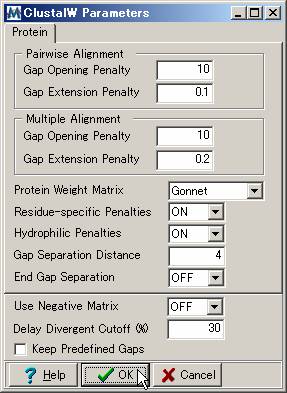
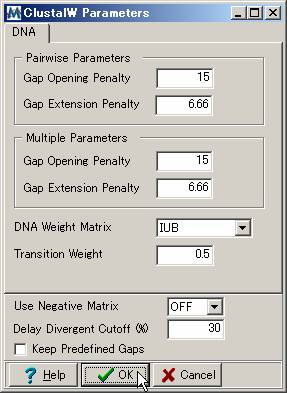
この条件で得られるアライメントのN末端領域(開始コドンのすぐ下流)のアライメント精度は通常よくない。アライメントの精度はDNA配列でも確認してみよう。
(4)
アライメントの最上行(灰色)をドラグし、最初の10アミノ酸座 (amino-acid sites) だけ選択、アライメントしてみる。
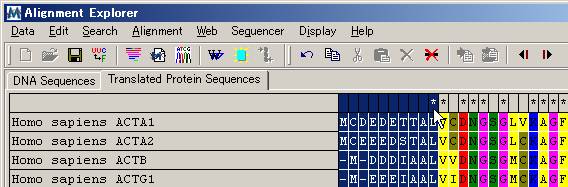
(5)
同じ領域について、「Gap Opening Penalty」(PairwiseとMultipleの2箇所)を「1」にしてアライメントしてみる。
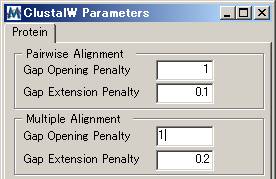
※理想的には領域毎に適切な「Gap
penalty」値を用いてアライメントを行うのがよい。
※構造・機能ドメインのような領域を区別するための情報がある場合、積極的に利用するとよい。
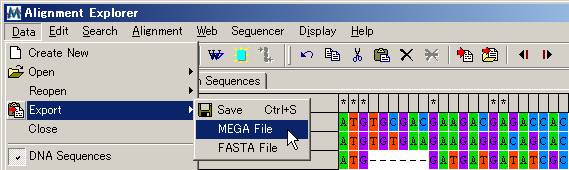
(6) 「DNA Sequences」タブをクリックし、DNA酸配列に戻した後、MEGA フォーマットでデータファイルを保存する。
【参考】
ドメイン毎の部分アライメント
タンパク質の機能ドメインや遺伝子のエクソン・イントロン構造などが分かっている場合、部分毎にアライメントする方が良いことが多い。例えば、全データのアライメントでは、エクソン部位とイントロン部位がアライメントされるようなエラーが起こる可能性があるが、を未然に防ぐことができる。
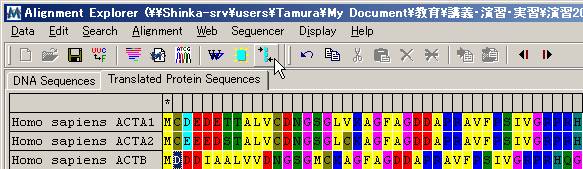
(1) ドメインの先頭をマークする(メニューの[Alignment]-[Mark/Unmark
Site]又はツールボタンをクリック、又は選択するサイトをCtrlキーを押しながらクリック)
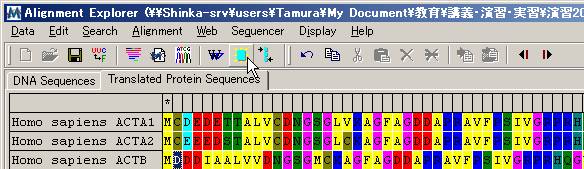

注: 検索([Search]-[Find Motif])を用いてマークするサイト周辺を見つけやすくできる。
(2) メニューの[Alignment]-[Align Marked
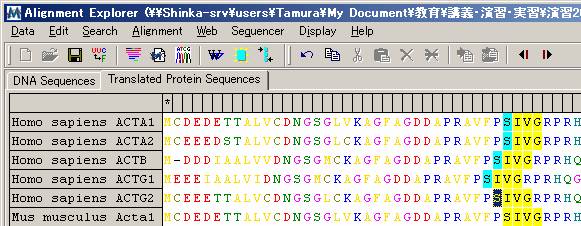
Sites]またはツールボタンのをクリックする(上図)とマークされたサイトがアライメントされる(下図)。
![]()
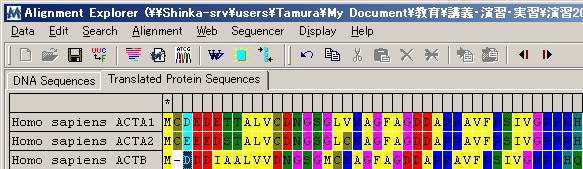
(3) 同様にドメインの最後をマークし、アライメントする。
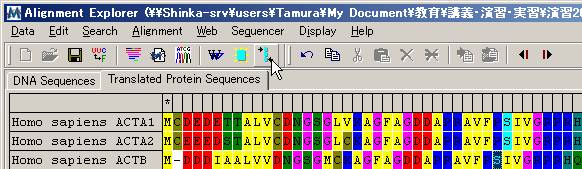
![]()
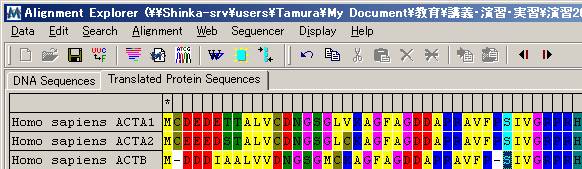
(4) ドメイン全体を選択し、アライメントする。
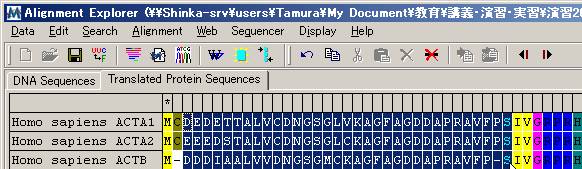
![]()